本章学习主要是针对如何挖掘数据之间的关联规则。如何离散化数据,使用Apriori关联规则算法,探索样本数据与预测结果直接的关联规则。
问题背景:假设你是一个超帅的医生,诊断肿瘤,你需要根据不同的病人症状来判断症状间的关系,规律,在不同阶段给病人开药,提高他活命的机会。生病的症状有很多种,彼此之间也是有关系的,比如因为你感冒了,所以发烧了,咳嗽了,流鼻涕了。所以我们需要分析不同症状之间的关系和规律,在病情恶化前尽量截断。
一、挖掘目标
1) 借助三阴乳腺癌的病理信息,挖掘患者的症状与中医证型之间的关联关系
2) 对截断治疗提供依据
二、数据抽取
1) 通过问卷获取患者个人信息
2) 通过问卷获取发病年龄、是否有各种症状等
如图是实际采集的数据:
三、数据预处理
1、 数据清洗:
由于是问卷调查,存在很多无效的问卷,所以根据数据是否有效进行筛选,筛选标准表如下:

2、 属性规约:(降维)
根据如下症状,去除与挖掘任务不相关的属性,选取6种证型得分和TNM

规约后的数据:
3、 数据变换:
- 1)属性构造
由于每种证型总分不相同,所以每种评分不是基于同一个标准,所以为了更好地反映证素的分布特征,采用证型系数代替证素得分,证型系数计算如下:
$证型系数=该证型得分/该证型总分$
ps:每个证型总得分为: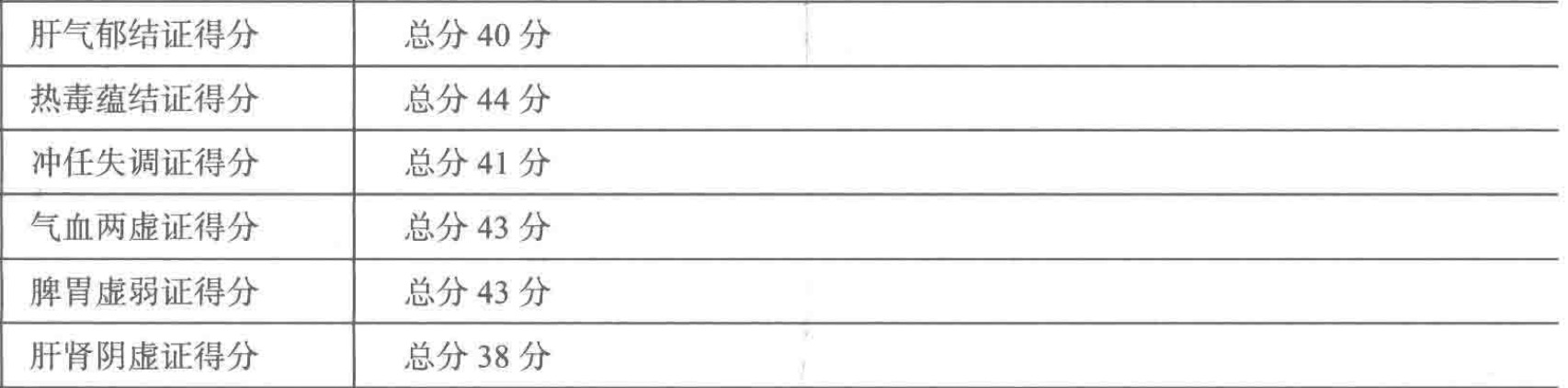
构造完毕的数据集如下: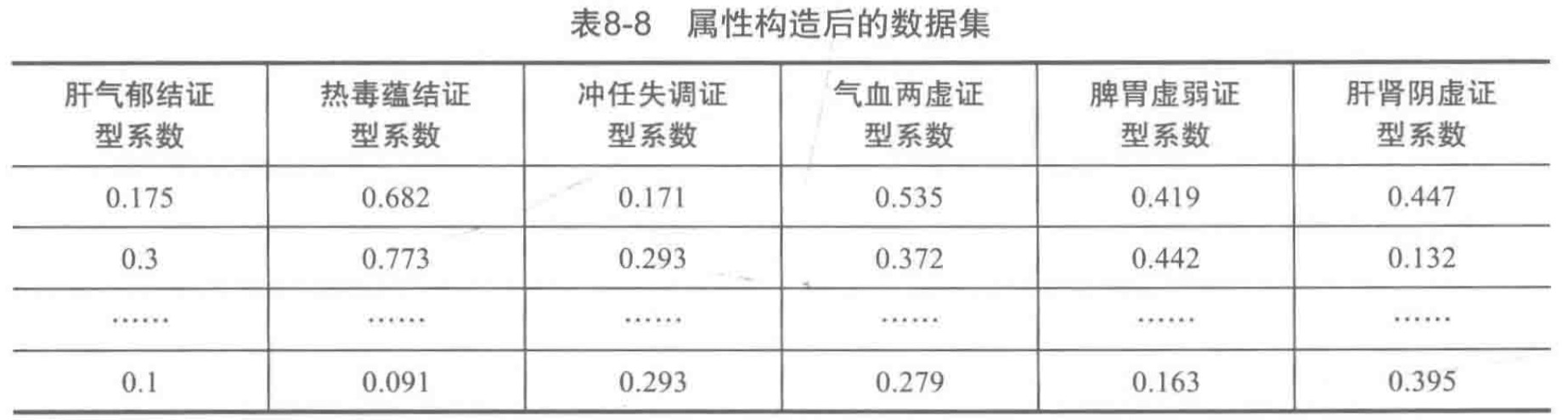
- 2)数据离散化
由于Apriori关联规则算法无法处理连续型数值变量,所以需要对数据进行离散化,这里采用聚类算法(请参考前面实验的K-Means算法详解)对证型系数进行离散化。
聚类离散化代码实现:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48#-*- coding:utf-8 -*-
'''
聚类离散化,最后的result的格式为:
1 2 3 4
A 0 0.178698 0.257724 0.351843
An 240 356.000000 281.000000 53.000000
即(0, 0.178698]有240个,(0.178698, 0.257724]有356个,依此类推。
'''
from __future__ import print_function
import pandas as pd
from sklearn.cluster import KMeans #导入K均值聚类算法
datafile='../data/data.xls' #待聚类的数据文件
processedfile='../tmp/data_processed.xls'#数据处理后文件
typelabel={u'肝气郁结证型系数':'A',u'热毒蕴结证型系数':'B',
u'冲任失调证型系数':'C',u'气血两虚证型系数':'D',
u'脾胃虚弱证型系数':'E',u'肝肾阴虚证型系数':'F'}
k=4 #需要进行的聚类类别数
#读取数据并进行聚类分析
data=pd.read_excel(datafile)#读取数据
keys=list(typelabel.keys())
result=pd.DataFrame()
if __name__ == '__main__':#判断是否主窗口运行,这句代码的作用比较神奇
#判断是否主窗口运行,如果是将代码保存为.py后运行,则需要这句,
for i in range(len(keys)):
#调用k-means算法,进行聚类离散化
print(u'正在进行"%s"的聚类...' %keys[i])
kmodel=KMeans(n_clusters=k,n_jobs=2)#n_jobs是并行数,一般等于CPU数较好
kmodel.fit(data[[keys[i]]].as_matrix())#训练模型
r1=pd.DataFrame(kmodel.cluster_centers_,columns=[typelabel[keys[i]]])
#聚类中心
r2=pd.Series(kmodel.labels_).value_counts()#分类统计
r2=pd.DataFrame(r2,columns=[typelabel[keys[i]]+'n'])#转为DataFrame,记录
#各个类别的数目
r=pd.concat([r1,r2],axis=1).sort_values(typelabel[keys[i]])#匹配聚类中心和类别数目,旧api:sort()
r.index=[1,2,3,4]
#rolling_mean()用来计算相邻2列的均值,一次作为边界点
r[typelabel[keys[i]]] = r[typelabel[keys[i]]].rolling(2).mean()#旧API:pd.rolling_mean(r[typelabel[keys[i]]],2)
r[typelabel[keys[i]]][1]=0.0#这两句代码将原来的聚类中心改为边界点
result=result.append(r.T)
result=result.sort_index()#以Index排序,即以A,B,C,D,E,F顺序排,旧api:sort()
result.to_excel(processedfile)
离散完的数据应如下:
四、模型构建
这里使用Arpiori算法(点击看该算法详解)
1、 由于Apriori算法时间较长,元数据比较大,鉴于这是实验,我们选用抽样的事务集,如下图:
2、 python使用apriori算法探寻关联规则(很遗憾,scikit-learn并未提供关联算法,所以这里的apriori算法是自己实现的库,请参考上面”Apriori算法”里该算法的实现):1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29##Apriori关联规则算法
#-*- coding:utf-8 -*-
from __future__ import print_function
import pandas as pd
from apriori import * #导入自行编写的高效的Apriori函数
import time #导入时间库用来计算用时
inputfile='../data/apriori.txt'#输入事务集文件
data=pd.read_csv(inputfile,header=None,dtype=object)
start=time.clock()#计时开始
print(u'\n转换原始数据至0-1矩阵...')
ct=lambda x:pd.Series(1,index=x[pd.notnull(x)])#转换0-1矩阵的过渡函数,即将标签数据转换为1
b=map(ct,data.as_matrix())#用map方式执行
data=pd.DataFrame(b).fillna(0)#实现矩阵转换,除了1外,其余为空,空值用0填充
end=time.clock()#计时结束
print(u'\n转换完毕,用时:%0.2f秒' %(end-start))
del b #删除中间变量b,节省内存
support = 0.06#最小支持度
confidence=0.75#最小置信度
ms='---' #连接符,默认'--',用来区分不同元素,如A--B.需要保证原始表格中不含有该字符
start=time.clock()#计时开始
print(u'\n开始搜索关键规则...')
find_rule(data,support,confidence,ms)
end=time.clock()#计时结束
print(u'\n搜索完成,用时:%0.2f秒' %(end-start))
执行结果:
3、 模型分析
根据上述运行结果,我们得出了5个关联规则,如A3–F–H4,它的意思是A3,F4=>H4,类似的, D2–F3–H4–A2的意思是D2,F3,H4=>A2。但是 ,并非所有关联规则都有意义的,我们只在乎那些 以 H为规则结果的规则(这里H就是我们想要预测的癌症TNM分期结果),也就是如下表的规则: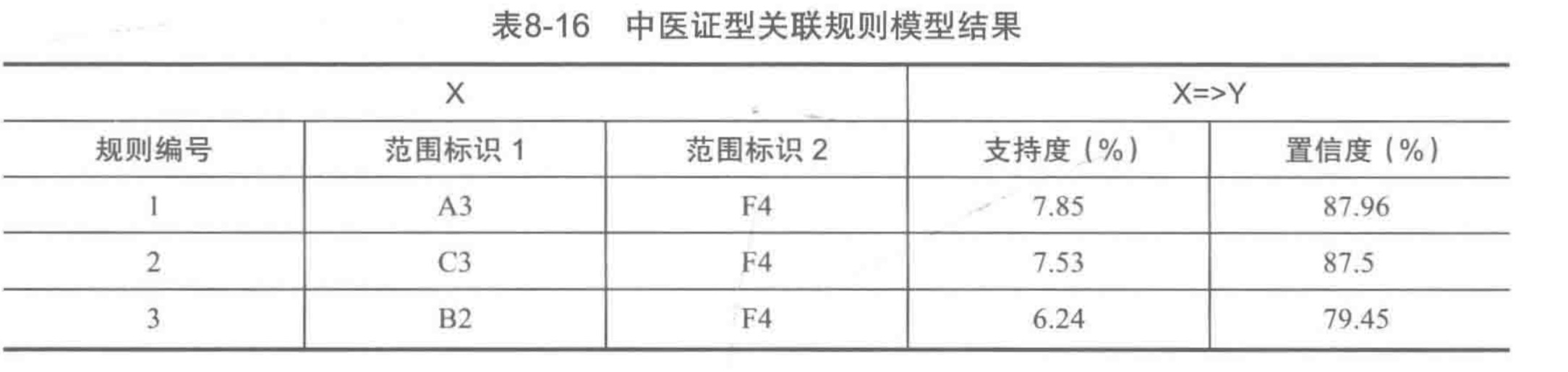
分析 上表可得到如下结论。
- 1) A3、F4=>H4支持度最大,达到了7.85%,置信度最大,达到了87.96%,说明肝气郁结证型系数在A3范围内,肝肾阴虚证型系数处于F4范围内,TNM分期诊断为H4期的可能性为87.96%,而这种情况发生的可能性为7.85%。
- 2) C3、F4=>H4支持度7.53%,置信度87.5%,说明冲任失调症型系数在C3范围内,肝肾阴虚证型系数处于F4范围内,TNM分期诊断为H4期的可能性为 87.5%,而这种情况发生的可能性为7.53%。
- 3) B2、F4=>H4支持度6.24%,置信度79.45%,说明热毒蕴结证型系数在B2范围内,肝肾阴虚证型系数处于F4范围内,TNM分期诊断为H4期的可能性为79.45%,而这种情况发生的可能性为6.24%。
综合以上分析,TNM分期为H4起价的癌症患者证型主要为肝肾阴虚证、热毒蕴结证、肝气郁结证和冲任失调,H4期患者肝肾阴虚和肝气郁结的临床表现较为突出,其置信度最大达到了87.96%。


